次世代シーケンス解析(MiSeq) MLST解析の解析事例
■MLST (Multi locus sequencing typing) 解析とは
複数の遺伝子領域 (通常 7 領域以上) の変異をパターン化して、菌のタイピングを行う方法です。16srRNA遺伝子解析では判別できなかった詳細な株の情報が得られます。従来のMLST解析は7つの遺伝子領域をPCR増幅して、サンガーシークエンスにより遺伝子領域ごとに配列決定を行い、パターンを決定する方法であり、各遺伝子領域のプライマー設計、PCRバイアス、サンプル数の増加による膨大な数のPCRなど煩雑な作業を含んでいました。これに対してNGSを利用したMLST解析はショットガンゲノムシークエンスにより得られた配列データを近縁種のゲノムにマッピングすることによって7つの遺伝子領域を同時に同定し菌のタイピングを行う方法であり、従来の方法に比べて迅速かつ高精度に行うことができると考えられます。
解析事例(自社データ)
単離株のゲノムと環境中の菌同定を想定した試料(単離株のゲノムに環境DNAに混合したもの)を用意し、タイピングを行いました。用意した株はBacillus subtilis subsp.subtilis str. 168(グラム陽性菌)、Pseudomonas protegens Pf-5(グラム陰性菌)で、これらのサンプルについてMiseqでゲノムシークエンス、タイピングを行いました。
◇ワークフロー
※CLC Microbial Genomics Moduleのツールを使用

◇結果
表1 アセンブル結果
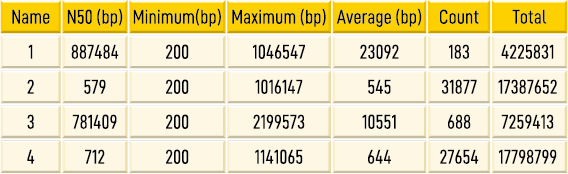
1: Bacillus subtilis subsp. subtilis str. 168
2: Bacillus subtilis subsp. subtilis str. 168 + soil DNA
3: Pseudomonas protegens Pf-5
4: Pseudomonas protegens Pf-5 + soi DNA
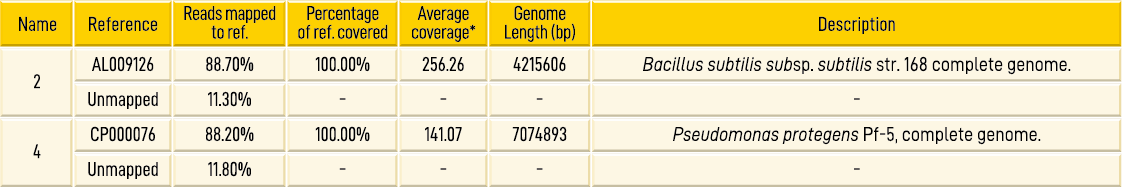
サンプル2と4に対してK-mer分布を用いて最も近縁な種を同定し、これらのゲノムをリファレンスとして、マッピングを行いました。
表2 マッピング結果
続いて、MLST解析に必要な7つの遺伝子領域を同定しました(glpF, ilvD, pta, purH, pycA, rpoD, tpiA)。図1はilvD: バリン、ロイシン、イソロイシンの合成経路に関わる遺伝子 (2,111 bp) へのマッピング結果を示しています。
図1 MLST解析対象の遺伝子領域へのマッピング結果
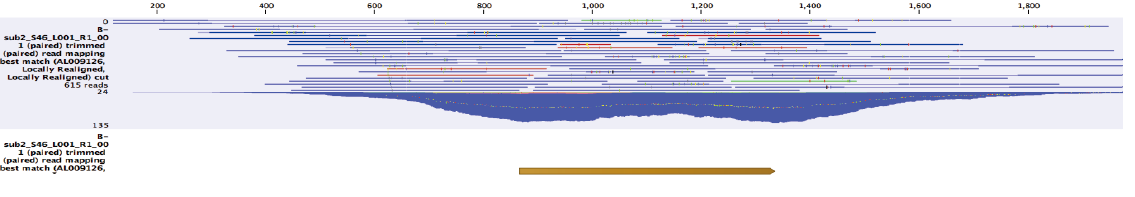
図2. NGS MLSTの解析結果

※詳細なTyping 結果はこちら
本手法での解析により、環境DNAに混在した株を同定することができました。このように優占種が大部分を占める環境には限定されますが、メタゲノムのような環境に対しても近縁種を推定してMLST解析を行うことが可能であるといえます。NGSを用いた本手法は今後、様々な分野で幅広く利用されることが期待されると考えられます。
サービスと価格
MLST解析はゲノムドラフト解析のオプションサービスとして承ります。
Sequence Typeのタイピング :+40,000円
※上記は解析費用です。サンプル調製費用やラン費用が別途かかります。

