GenomeCraft 使用例
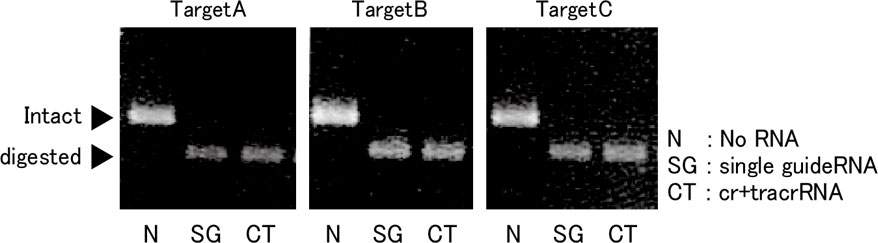
● In vitro digestion assay
Cas9タンパク質とsgRNAまたはcrRNA + tracrRNA、基質DNA断片を混合しDNA切断実験を行った。37 °Cにて1時間インキュベート後、アガロースゲル電気泳動を行った。
● In vivo Genome Editing
Cas9タンパク質とsgRNAまたはcrRNA + tracrRNAを混合し、メダカ胚にインジェクションした。4日胚まで発生させた各個体のゲノムDNAからターゲット領域をPCR増幅してheteroduplex mobility assaysにて変異の検出を行った。下図の各レーンごとに1個体分の結果を示している。
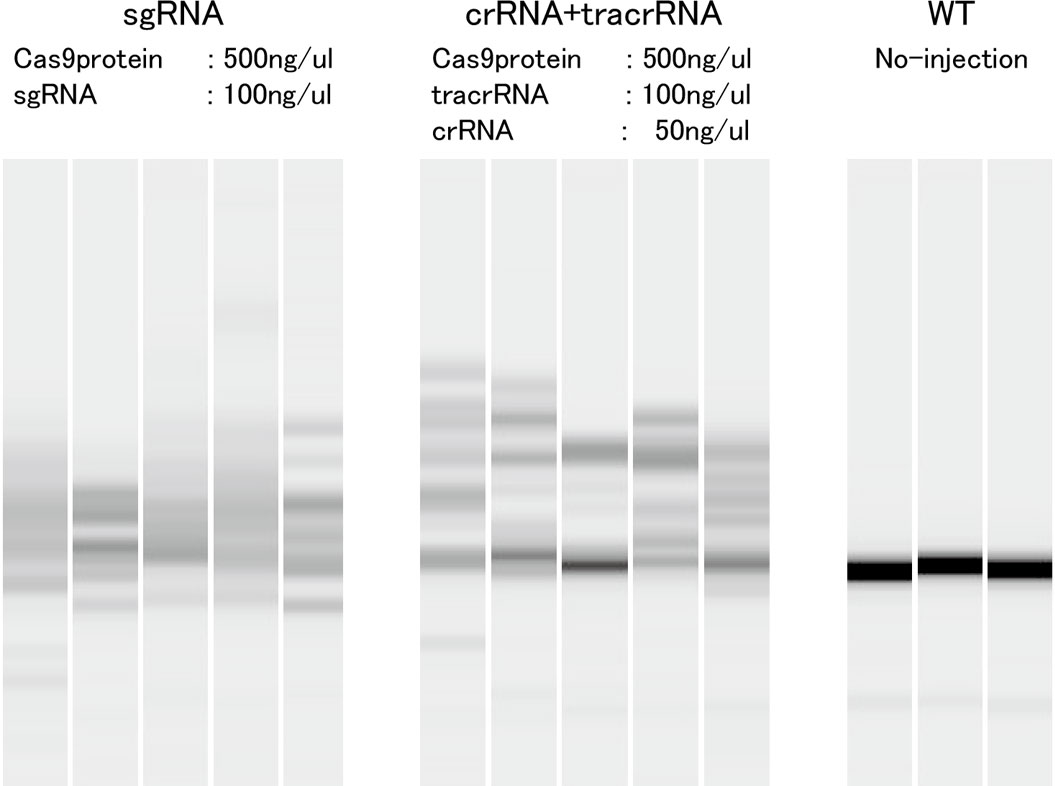
sgRNA、crRNA + tracrRNAともに変異の存在を示す複数のバンドが観察され、高効率な変異導入ツールであることが確認された。
データ提供 京都大学 農学研究科 応用生物科学専攻 海洋生物機能学分野 木下 政人 様
※ sgRNAは木下ラボで合成し、crRNAとtracrRNAはファスマック提供。
● ダブルノックアウト (ゼブラフィッシュ)
Cas9タンパク質と2つの遺伝子 (spns2: 心臓形成に関与、tyr: 色素形成に関与)に対するsgRNAまたはcrRNA + tracrRNAを混合し、ゼブラフィッシュ胚 (1細胞~2細胞期)にインジェクションして、発生の様子を観察した。
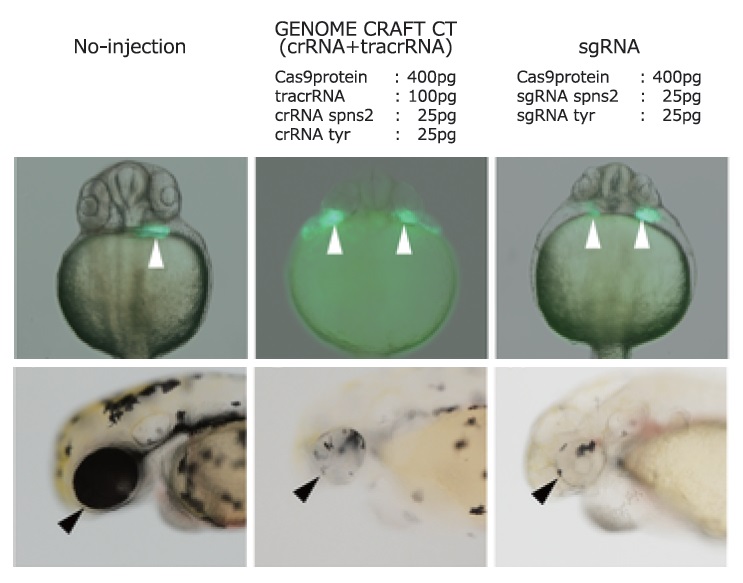
受精1日後 (上段)にGFPで染色された心臓の形成異常 (白矢印)が、2日後 (下段)には目の色素形成異常 (黒矢印)が観察された。これらはそれぞれspns2欠損、tyr欠損の表現型と同一である。またシークエンス解析により各遺伝子への変異導入が確認された。
データ提供 山梨大学大学院 総合研究部 発生生物学教室 川原 敦雄 様※ 下記文献より改変
● 遺伝子ノックイン (マウス)
Cas9タンパク質とActb遺伝子対するsgRNAまたはcrRNA + tracrRNA、ターゲティングドナーベクターを混合し、マウス受精卵に注入してノックインマウス (約2.5 kbpの遺伝子カセット導入)の作成を試みた。注入された受精卵から生まれた仔マウスに対して、PCRでWTアリル、ノックイン (KI)アリルの有無調べた。
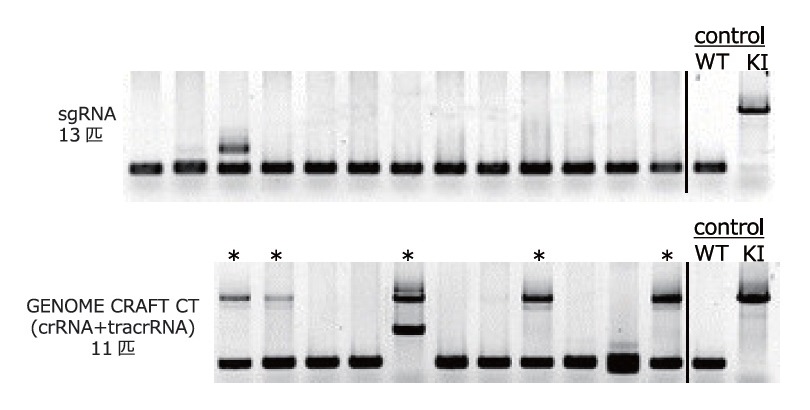
sgRNAを用いた場合には仔マウス13匹中にKIマウスは得られなかったが、crRNA + tracrRNAを用いた場合では11匹中にKIマウス5匹 (*)を得ることができ、ゲノムの意図した場所へ、正確で高率に遺伝子挿入できることが確認された。
データ提供 東京医科歯科大学 難治疾患研究所 分子神経科学分野 相田 知海 様, 田中 光一 様※ 下記文献より改変

