DNBSEQによる解析
MGI社のDNBSEQを用いたローコストな解析をご提案いたします。
DNBSEQとは、MGI社が提供するショートリードのシーケンサーです。1ランあたり最大640 Gのシーケンスデータのアウトプットを実現し、イルミナ社のシーケンサーと変わらないデータクオリティとなっております。
弊社はDNBSEQの中でも中出力機器であるDNBSEQ-G400を導入しました。DNBSEQの導入により、大規模なシーケンシングが可能となり、真核生物に向けたサービスを幅広く提案していきます。
またRNA-seq(トランスクリプトーム)やゲノムドラフトなど既存のサービスもDNBSEQに置き換えれば安価、データ増量、多検体解析を実現できます。なお、全ての解析は日本国内の自社で実施しておりますので、情報保護の観点からもご安心いただき、ご活用ください。

価格
納期は共有ランが25営業日、専有ランは20営業日が目安となります。
(1)DNBSEQでのラン
| 作業内容 | 価格 | データ量 *理論値 |
リード数(pair) *理論値 |
|---|---|---|---|
| 1レーン専有ラン(2×150 bp) | 220,000円 | 82 G | 275 M |
| 共有ラン(2×150 bp) | 25,000円 | 6 G | 20 M |
| 1レーン専有ラン(2×300 bp) | 500,000円 | 90 G | 150 M |
※Unique Dual Indexを使用し、ライブラリ調製をおこなっております。
※原則として、共有ラン1件につき、8 Index(8サンプル)までの受け入れとなります。
専有解析の場合、弊社でご用意があるindex数は96までとなります。
※また弊社ではilluminaライブラリのコンバージョンによる調製をおこなっております。
コンバージョンの際、5 cycle~のPCRを実施しております。
(2)サンプル調製費用、クオリティチェック費用
| 項目 | 価格 | |
|---|---|---|
| QC(quality control)作業[必須] | 濃度・品質チェック | 16サンプルまで 30,000円 |
| ゲノムショットガン | DNBSEQ用ライブラリ調製 | 15,000円/サンプル |
| RNA-seq | DNBSEQ用ライブラリ調製 | 25,000円/サンプル |
| アンプリコン*1 | DNBSEQ用ライブラリ調製 | 40,000円/8サンプル |
| RNA抽出*2 | 市販のキットを用いて抽出 | ご相談ください |
| DNA抽出*2 | 市販のキットを用いて抽出 | ご相談ください |
| illuminaライブラリの受け入れ | お客様で調製*3 | ご相談ください |
*1 アンプリコンライブラリをランする場合には1:1程度の割合で PhiX ライブラリーを添加します。
出力データについてもカタログスペックの50%以下となりますので、ご承知おきください。
*2 弊社でノウハウのないものはお断りさせていただく場合があります。
*3 お送りいただくライブラリ濃度は5 ng/μl・40 μl以上を推奨いたします。
ご依頼の流れ
①お見積り依頼
までご要望の解析内容をお知らせください。
②ご依頼
弊社Webログインサイトより事前にサンプル情報等をご登録ください。
③サンプル送付
ログインサイト登録後に自動配信されるメールを印刷し、サンプルにご同梱ください。
④弊社での受け入れ
ご同封いただいたサンプルシートとサンプルを照合し、注文受付いたします。
⑤解析、納品
解析例
・全ゲノムシーケンス
ラージサイズのゲノム解析をベースにマッピングや変異解析(SNP)などが指定可能です。真核生物におすすめです。
【参考価格】
真核ゲノム1サンプル(カバレッジ30)、アセンブルとコンティグを含んだ場合:305,000円(税抜き)
*内訳→QC費用:30,000円+ライブラリ調製費用:15,000円+1レーン専有(2×150 bp):220,000円+アセンブルとコンティグ作成:40,000円
・RNA seq解析(トランスクリプト―ム)
発現量算出(TPMやFPKM)に加えて、発現比較解析やヒートマップ作成などを行います。DNBSEQであればお求めやすい価格で多検体の解析を実現できます。
【参考価格】
8サンプル、TPMによる発現量算出の場合:360,000円(税抜き、1サンプルあたり¥45,000)
*RNAseqパッケージ価格:QC費用、ライブラリ調製費用、ラン費用(20Mリード/サンプル)、発現量算出込み
イルミナ社製Next-seqとDNBSEQの比較(RNA-seq)
ヒト培養細胞から抽出したTotal-RNAを用いて、RNA-seq用のライブラリ調製をそれぞれのプラットフォームの推奨に従って実施し(2反復)、ランを行いました。そして取得したデータについて遺伝子発現量の算出を行い、プラットフォーム間での発現量比較を行いました。
シーケンスの統計情報 Next-seq:2×75 bp /DNBSEQ: 2×100 bp
| Sample name | Number of reads | Paired, mapped % | Paired, not mapped % | Mapped to genes % | Mapped to intergenic % | mRNA (%) |
|---|---|---|---|---|---|---|
| DNBSEQ_1 | 45,722,649 | 96.56 | 0.83 | 97.75 | 2.25 | 100 |
| DNBSEQ_2 | 56,856,171 | 96.51 | 0.84 | 97.79 | 2.21 | 100 |
| Next-seq_1 | 30,212,140 | 93.14 | 2.92 | 98.66 | 1.34 | 100 |
| Next-seq_2 | 24,473,703 | 92.78 | 2.95 | 98.69 | 1.31 | 100 |
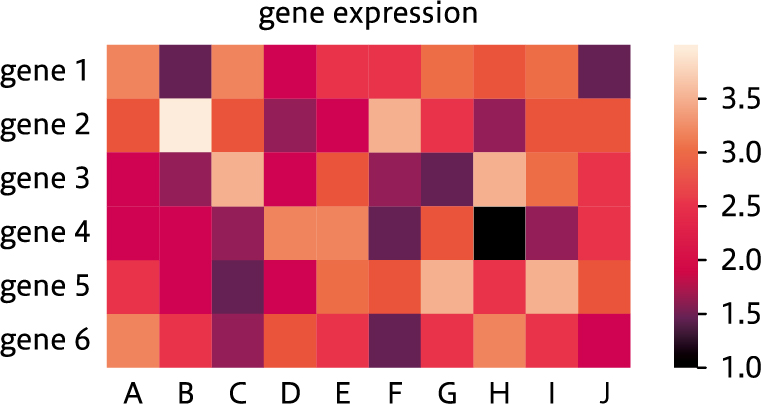
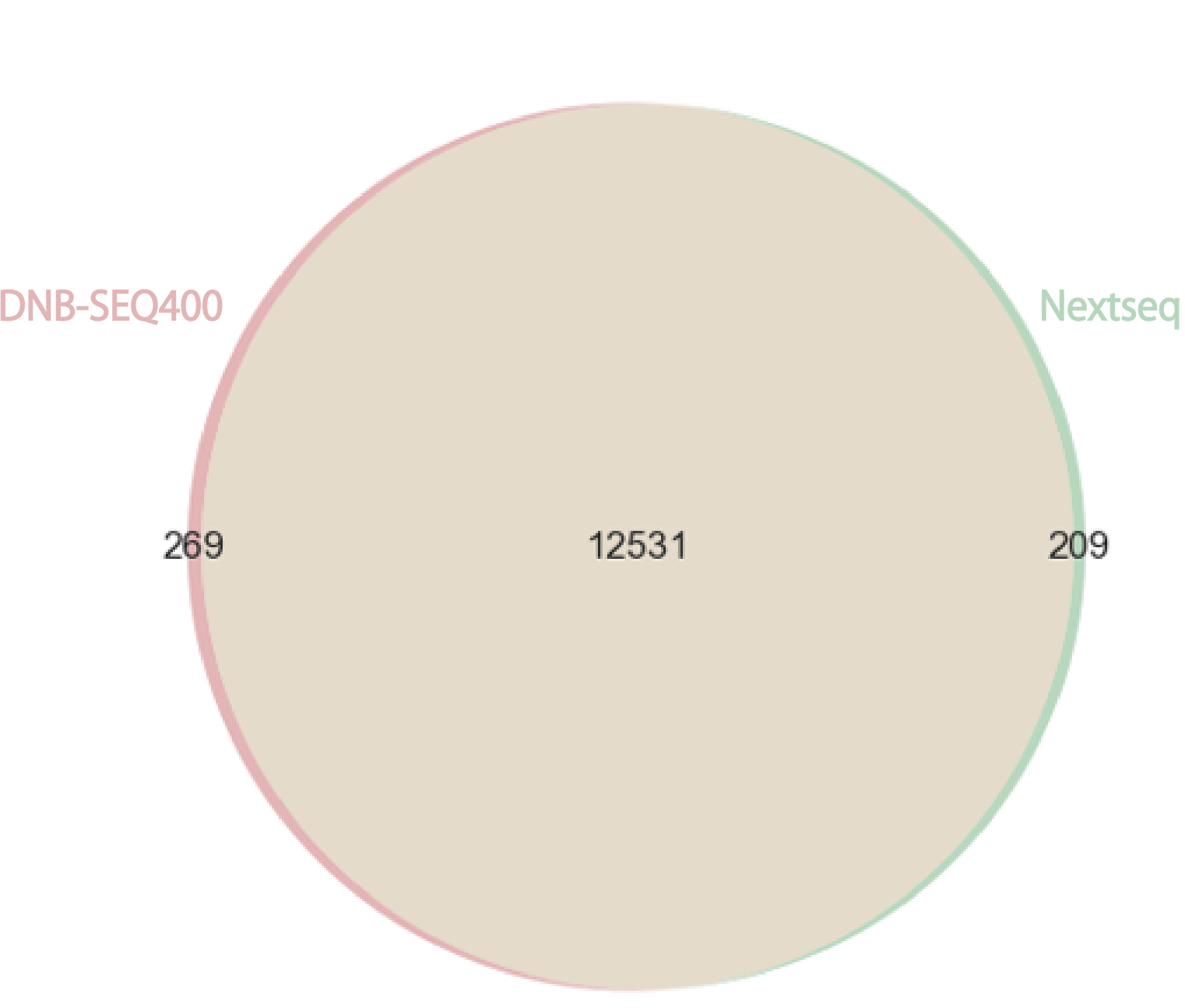
遺伝子発現の相関を解析したところ、両プラットフォーム間で高い相関係数を示しました(Fig.1)。またFig.2では各プラットフォームで検出した遺伝子の違いをベン図で示しています。結果、非常にきれいに重なっており、検出した総遺伝子の内、96 %以上が共通して検出された遺伝子でした。
Fig.1 遺伝子発現の相関ヒートマップ
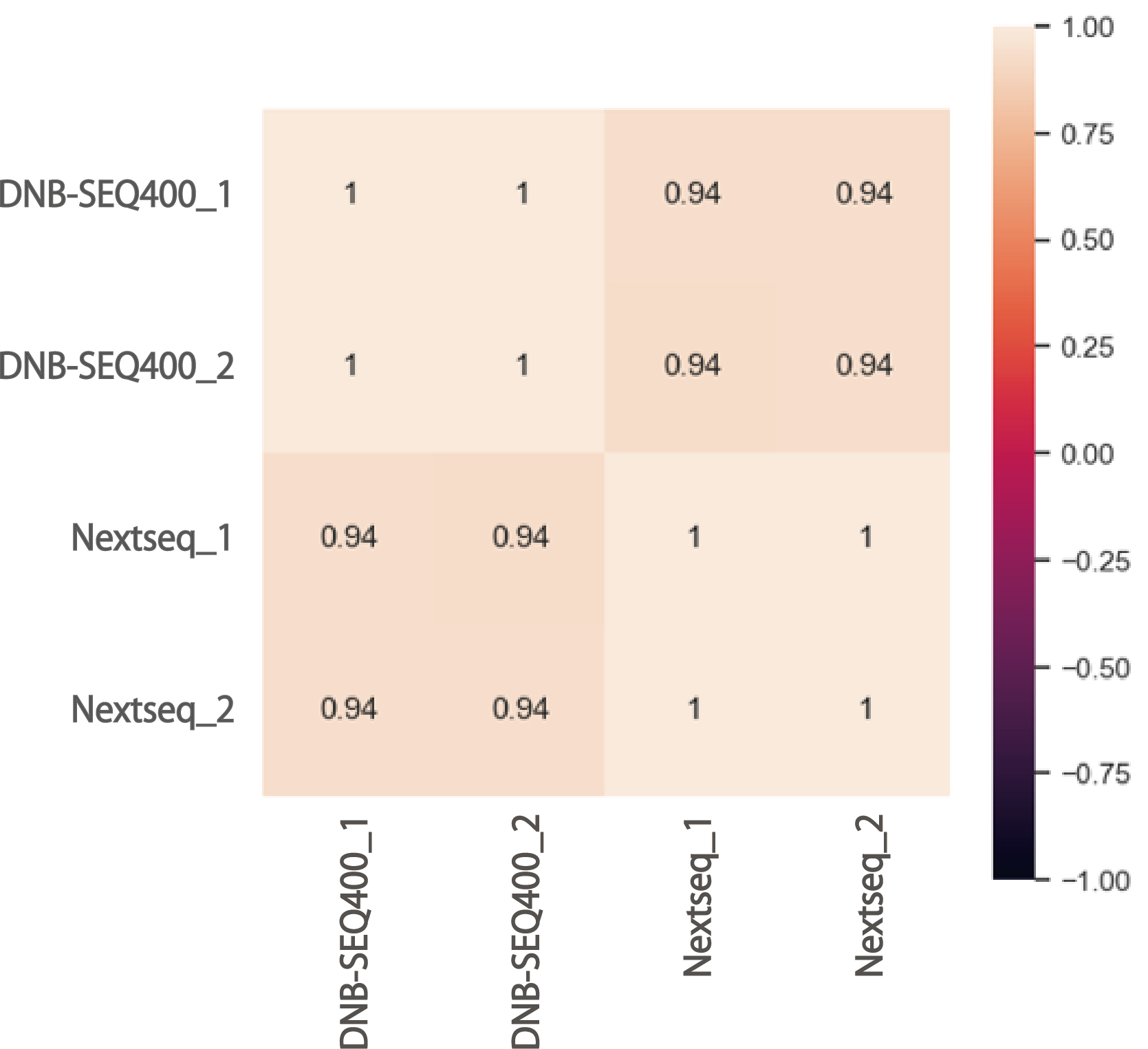
Fig.2 ベン図での遺伝子の比較(TPM1以上)
DNBSEQとイルミナシーケンサー、どちらがよいか迷ったら
例えばMiseqはDNBSEQよりも読み取り長の面で優れており、弊社の主力サービスであるアンプリコン解析では欠かせません。またゲノムドラフトでもロングリードの方がコンティグ形成に寄与します。ゲノムサイズが小さいもの(ウイルスや原核生物など)の場合にはMiseqがおすすめです。お客様のご用途とご要望に応じてこちらで判断し、最適なプランを提案させていただきますのでご安心ください。
【問い合わせ】
株式会社ファスマック 次世代シーケンス解析サービス
〒243-0021 神奈川県厚木市岡田3088 ケーオービルA棟4階
TELL:046-281-9909/FAX 046-281-9931


