次世代シーケンス解析(MiSeq) ジェノタイピングの解析事例
ゲノム編集技術は自由な遺伝子改変を可能にするパワフルなツールですが、改変個体を選抜する上で、遺伝型解析(ジェノタイピング)は重要な工程です。F1 世代以降でジェノタイピングを行い、改変個体を獲得するのが一般的です。理由は改変後のF0 世代は、モザイク(同一個体中に遺伝子型の異なる細胞が混在する)になる傾向が強いためです。しかしながら、次世代シーケンスにより、F0世代でのアリル頻度を評価することで、交配に用いるべき個体選抜を行うことができます。また、培養細胞のゲノム編集実験では1度の実験で多様な変異を持つ細胞集団が得られ、実験条件によって変異の様子が異なります。次世代シーケンスなら細胞集団の変異を詳細に評価することが出来ます。
解析事例:CrispRvariantを用いた培養細胞のジェノタイピング
データ提供:安田秀世様(Konkuk University,Korea)
関連文献:J Genet Genomics.2016 Dec 20;43(12):705-708
A highly efficient method for enriching TALEN or CRISPR/Cas9-edited mutant cells.
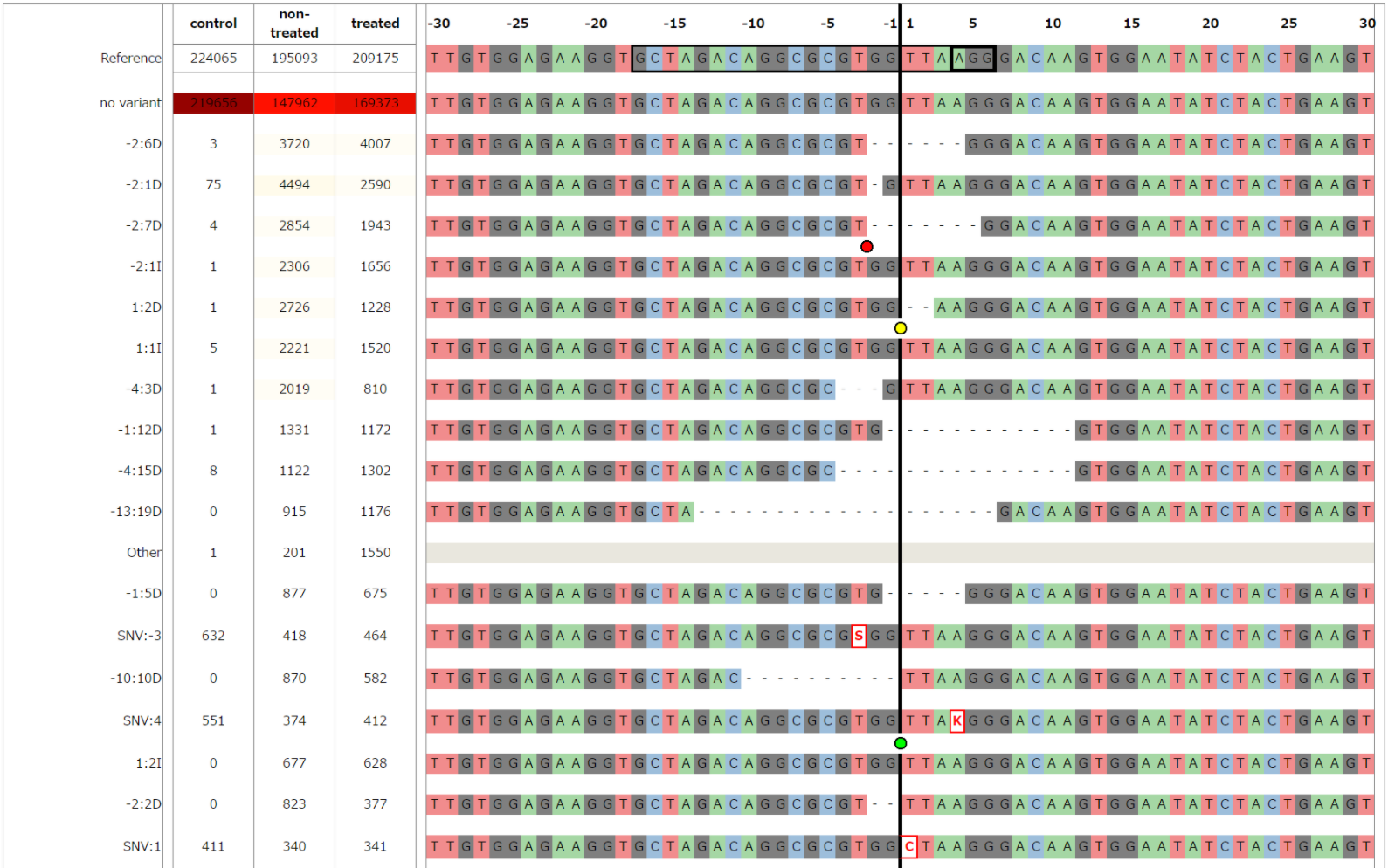
CRISPR/Casでゲノム編集した培養細胞株からDNA抽出後、ターゲットアンプリコンシーケンスをMiseqにて実施。得られたデータをCrispRvariantにて解析しました。次世代シーケンスを利用することでより高い解像度でジェノタイピングを行うことが可能です。
・CrispRVariant(改変部位の変異配列やアリル頻度を分かりやすく示すことが出来ます。)
・ジェノタイピングテーブル(エクセル形式)【ダウンロード】(39kb)
※解読長の関係上、CrispRvariantはノックアウト、もしくはノックインの前後で想定されるターゲットアンプリコンサイズが500bp未満の場合に利用できます。
また、キャピラリーシーケンサーを用いたフラグメント解析(サイジング)により、個体のindel判定も可能と考えております。お気軽にご相談下さい。
※F1個体での解析を推奨いたします。
また、弊社ではガイドRNAやCas9タンパクといったCRISPR/Casのゲノム編集に使用するツールの販売を行っております。また人工遺伝子サービスやオリゴDNA合成サービスはノックイン用ドナーDNAの作製にご利用いただけます。併せてご検討下さい。