次世代シーケンス解析(MiSeq) 生菌微生物群集構造解析の解析事例
選択的膜透過性色素EMA*(EMA:ethidium monoazide)を用いた群集構造解析(自社データ)
*タカラ社のものを使用
EMAが死菌由来DNAを修飾し、修飾を受けたDNAがPCR増幅できない状態となることを利用して、生菌由来DNAを選択的に検出する方法です。
大腸菌が優占した環境サンプル (培養した大腸菌 (K13由来株)をサンプルに添加) について、熱処理して殺菌したサンプルと処理しない (生きている) サンプルを調整し、EMA処理 (グラム陰性菌対象) を行った(比較対象として未処理のサンプルも用意)。その後、各サンプルに対してDNA抽出を行い、16Sr RNA遺伝子アンプリコン解析を実施した。
◇ワークフロー

※DNA抽出については、お客様のサンプル状況に合わせて適切なキットを使用しております。
◇結果
EMA処理をしていないサンプルに関してはライブラリーのDNA濃度及び群集構造、両方の結果に差異が見られなかった一方でEMA処理したサンプルについては熱処理をしたサンプルのライブラリー濃度はPCR増幅が阻害され濃度は低く(表1)、さらに群集構造解析では優占化した大腸菌が死滅したことによって熱処理したサンプルでは大腸菌はほとんど検出されませんでした(図1)。これはEMAによってDNAが不活性化されPCRの増幅が阻害されたことが予想されます。このことからEMAを用いた手法は生菌群集を解析することが可能であることが示されました。
図1 EMAを用いた微生物群集構造解析
※環境サンプル : 九州地域の堆積土
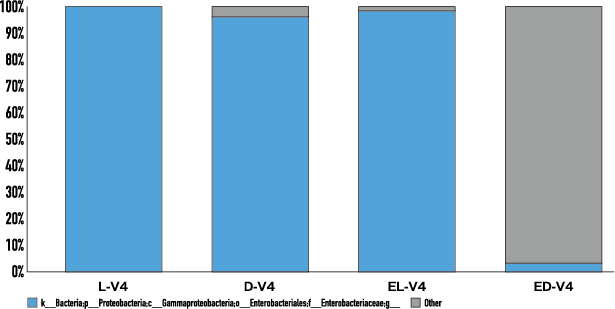
表1 ライブラリーのDNA濃度
| Qubit | |
|---|---|
| Name | library(ng/ul) |
| L | 34.9 |
| D | 19.4 |
| EL | 17.9 |
| ED | 1.06 |
L :熱処理していないサンプル (生菌サンプル)
D :熱処理して死滅させたサンプル (死菌サンプル)
EL:EMA処理した生菌サンプル
ED:EMA処理した死菌サンプル
RNAからの逆転写cDNAによる群集構造解析(自社データ)
環境サンプル中のRNAを解析することにより、実際に活動している微生物を特定する方法です。
培養サンプルと環境サンプルからRNA抽出を行い、さらにDNA除去のために精製処理を行った後、逆転写反応を行った。その際、逆転写を行っていないサンプルをネガティブコントロールとした。そして16Sr RNA遺伝子をターゲットとしたアンプコンライブラリーを作製した。
◇ワークフロー

上のワークフローにおいて各ステップ間でQubit測定を実施した。サンプルとして、培養株 Burkholderia multivorans、環境サンプル : 九州地域の堆積土を用いた。Aは逆転写を行い、Bは逆転写を行っていないサンプル(NTC)である。
表2 抽出精製後やライブラリー調製後など各ステップにおけるQubitによるDNA及びRNAの濃度
| Qubit① | Qubit② | Qubit③ | |||
|---|---|---|---|---|---|
| Name | RNA | DNA | RNA | DNA | library |
| 培養株RNA_A | 11.1 | High | low | low | 43.8 |
| 培養株RNA_B | 11.1 | High | low | low | low |
| 培養株RNA2_A | 38.5 | 36.3 | low | low | 53 |
| 培養株RNA2_B | 38.5 | 36.3 | low | low | low |
| 環境サンプル1_RNA1_A | 14.8 | 22.6 | 2.91 | low | 48 |
| 環境サンプル1_RNA1_B | 14.8 | 22.6 | 2.91 | low | 0.194 |
| 環境サンプル1_RNA2_A | 17.8 | 26 | 2.82 | low | 45.2 |
| 環境サンプル1_RNA2_B | 17.8 | 26 | 2.82 | low | low |
| 環境サンプル3_RNA2_A | 17.3 | 26.4 | 3.64 | low | 46.2 |
| 環境サンプル3_RNA2_B | 17.3 | 26.4 | 3.64 | low | low |
◇結果
RNA抽出精製後、ライブラリ調製後の濃度が、多くの解析サンプルにおいて良好な値を示しました(表2)また、調製したライブラリがDNA由来でないことを確かめるため、比較対象として逆転写反応を行わずPCR増幅したものをNTCとしましたが、こちらの濃度はごくわずか、もしくは検出限界以下でした(表2、B)。このことは、今回の結果がサンプルDNAの由来ではなくRNAの群集構造であることを裏付けております(図2) 。今回の検証から、本ワークフローが高品質なRNA及びライブラリーを調製するのに有効であることが示唆されました。
図2. cDNAの微生物群集構造解析
■培養株 ※RNAは2反復で実施
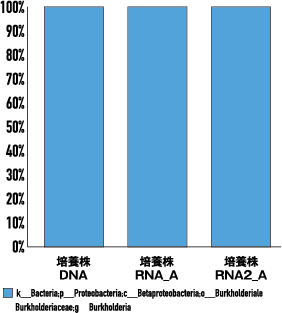
■環境サンプル: 九州地域の堆積土 ※RNAは3反復で実施

EMAやRNAによる手法は、DNA群集構造解析では得られない実際の環境の生菌の群集構造をアプローチする非常に重要なツールといえます。
EMAによる解析手法では、菌数が少ない環境においても適用可能となります。また、RNAによる解析手法では、菌数が多く、DNAの収量が多い環境でないと、環境サンプルからRNAを得ることが非常に難しいと予想されますが、EMAに比べ菌種による偏りはないと考えられます。これらの手法は相補的な関係をもつと考えられ、組み合わせて利用することもお勧めいたします。
サービスと価格
生菌微生物群集構造解析は、ターゲットアンプリコン解析のオプションで承っております。
| 内容 | 価格 | |
|---|---|---|
| RNAからの逆転写による アンプリコン解析 | RNA抽出から | +35,000円/サンプル |
| 逆転写PCRから | +20,000円/サンプル | |
| EMAによるアンプリコン解析 (EMA処理→DNA抽出→PCR) | +35,000円/サンプル | |
※上記はライブラリ調製の費用です。Miseq費用(QC/ラン/解析)が別途かかります。

